3.经典语义分割网络U-Net论文解读
欢迎访问个人网络日志🌹🌹知行空间🌹🌹
文章目录
- 1.基础介绍
- 2.`Overlap-tile strategy`
- 3.网络模型
- 4.损失函数
- 参考资料
1.基础介绍
论文:U-Net: Convolutional Networks for Biomedical Image Segmentation
工程:https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/
这是德国弗赖堡大学2015年05月份发表的论文,最早接触U-Net是2017年的时候,现在再回过头来看下这些经典全卷积语义分割网络。截至2023年03月份,这篇文章的引用已经达到了57800多次,从这里可以看出这篇文章在语义分割方向的地位,也能看出AI+CV的热度之高。
U-Net网络的提出是为了对医学领域细胞电镜的图像进行分割,这个任务特殊之处在于医学图像获取比较困难,因此只能从小样本数据中学习。作者通过对训练数据进行增强来学习,医学细胞图像多样性没有那么多,但更多的是旋转,尺度,形变和亮度这些。作者提出的端到端训练的全卷积网络包括特征提取的压缩路径和上采样的扩展路径,比较早的采用了这种编解码结构的模型。医学图像中特殊的地方还有一处就是,细胞虽然属于同个类别,但不是同个细胞时还需要把细胞间的间隔背景给识别出来,为此作者提出了一种加权的损失函数,增大了间隔背景的损失权重,更利于模型的学习。
总结这篇文章的主要工作有以下几点:
- 包含压缩路径和扩展路径的编解码结构的全卷积分割模型,实现了端到端训练
- 提出了一种加权损失函数,更利于学习个体之间的间隔背景
- 基于数据增强的小样本学习及一种利于边缘像素预测的
Overlap-tile strategy
2.Overlap-tile strategy
为了避免在分割的边沿产生了类似于padding的黑边,文中作者提出了Overlap-tile strategy,原理就是沿着边沿取图像的一部分,然后将其沿着边镜像,通过这中方式将原图的size进行扩大,避免在训练时对图像原图的边沿进行填充。
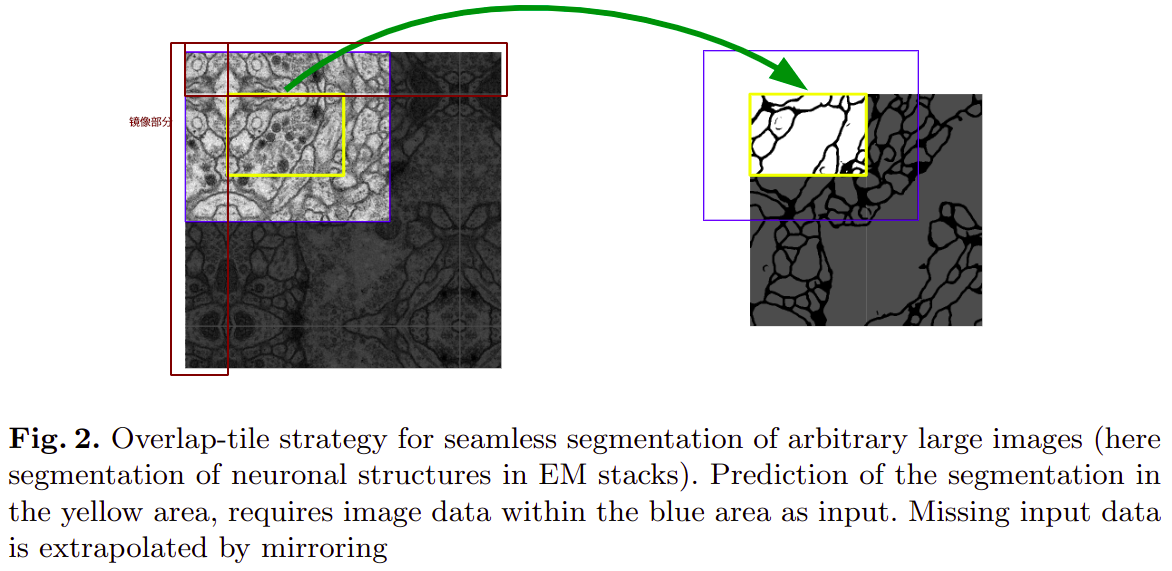
如上图,原来输入图的大小是388,左右上下取原图像上92像素镜像扩展得到输入图像的大小为572。
3.网络模型
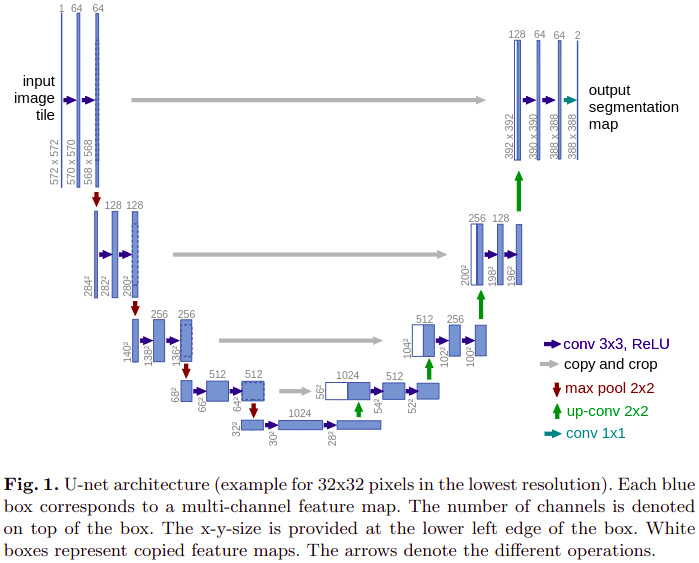
与之前介绍的FCN中不同,这里特征融合使用的是,低层特征图和高层特征图直接在通道方向上concatenate后得到。
压缩路径,2个uppadded的3x3卷积层, 后跟ReLU和2x2的stride=2的最大值池化层
每下采样一次,卷积的通道数翻倍
扩展路径,2x2的转置卷积,2倍上采样,通道数减半,与压缩路径中对应大小的feature map concatenate,再使用两个conv3卷积层使通道数减半,最后使用1x1的卷积在通道上输出每个像素位置所属类别的结果。
论文中,只需要分割出是否是细胞,因此最后卷积输出的通道数即类别数是2.
4.损失函数
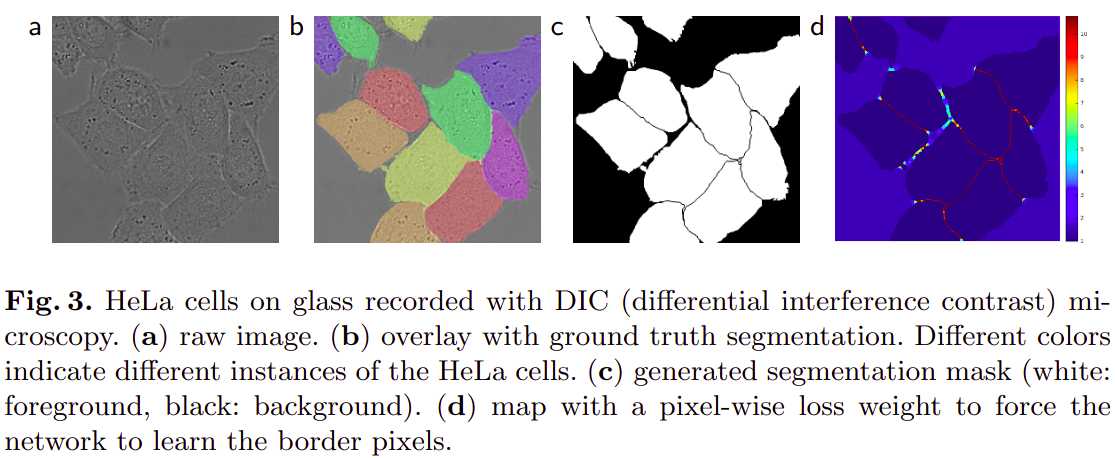
如上图中b,cb,cb,c,细胞之间的间隙背景非常小,但又非常重要,如果不采取特殊的方式,小部分的背景将很难分割出来,为此,作者提出了加权损失函数。
最后一层卷积的输出在通道方向上使用SOFTMAX函数可以得到类别概率图,
pk(x)=exp(ak(x))∑k′Kexp(ak′(x))p_k(x) = \frac{exp(a_k(x))}{\sum\limits_{k'}^Kexp(a_{k'}(x))} pk(x)=k′∑Kexp(ak′(x))exp(ak(x))
ak(x)a_k(x)ak(x)表示在卷积特征图位置$x\in\Omega 通道通道通道k上经过激活函数后的输出。上经过激活函数后的输出。上经过激活函数后的输出。\Omega \subset \mathbb{Z}^2,其中,其中,其中\mathbb{Z}表示自然数。表示自然数。表示自然数。K是总的类别数。是总的类别数。是总的类别数。p_k(x)表示的是softmax后类别为表示的是softmax后类别为表示的是softmax后类别为k$的概率。
交叉熵损失函数的定义:
E=∑x∈Ωw(x)log(pl(x)(x))E = \sum\limits_{x\in\Omega}{w(x)log(p_{\mathscr{l}(x)}(x))} E=x∈Ω∑w(x)log(pl(x)(x))
l:Ω⟶{1,..,K}\mathscr{l}:\Omega\longrightarrow \{1,..,K\}l:Ω⟶{1,..,K}是每个像素的真实标签,w∈Ω→Rw\in \Omega\rightarrow\mathbb{R}w∈Ω→R是权重图,为了指定某些像素在训练时更重要,这里是细胞间隔背景。这个权重图是根据标签分割图生成的。
w(x)=wc(x)+w0⋅exp(−(d1(x)+d2(x))22σ2)w(x) = w_c(x) + w_0\cdot exp(-\frac{(d_1(x)+d_2(x))^2}{2\sigma^2}) w(x)=wc(x)+w0⋅exp(−2σ2(d1(x)+d2(x))2)
wc:Ω→Rw_c:\Omega\rightarrow \mathbb{R}wc:Ω→R表示wcw_cwc是与标签分割图大小相同的实数权重图。d1:Ω→Rd_1:\Omega\rightarrow \mathbb{R}d1:Ω→R表示当前位置距离最近细胞边沿的像素距离,d2:Ω→Rd_2:\Omega\rightarrow \mathbb{R}d2:Ω→R表示当前位置距离次最近细胞边沿的像素距离,在论文中w0=10,σ≈5w_0=10,\sigma\approx 5w0=10,σ≈5。
获取权重图的一个pytorch实现的代码示例如下:
class CellDataset(Dataset):...def _get_boundary_weight(self, target, w0=10, sigma=5):"""This implementation is very computationally intensive!about 30 minutes per 512x512 image"""print('Calculating boundary weight...')n, H, W = target.shapeweight = torch.zeros(n, H, W)ix, iy = np.meshgrid(np.arange(H), np.arange(W))ix, iy = np.c_[ix.ravel(), iy.ravel()].Tfor i, t in enumerate(tqdm(target)):boundary = find_boundaries(t, mode='inner')bound_x, bound_y = np.where(boundary is True)# broadcast boundary x pixeldx = (ix.reshape(1, -1) - bound_x.reshape(-1, 1)) ** 2dy = (iy.reshape(1, -1) - bound_y.reshape(-1, 1)) ** 2d = dx + dy# distance to 2 closest cellsd2 = np.sqrt(np.partition(d, 2, axis=0)[:2, ])dsum = d2.sum(0).reshape(H, W)weight[i] = torch.Tensor(w0 * np.exp(-dsum**2 / (2 * sigma**2)))return
代码引用自2
欢迎访问个人网络日志🌹🌹知行空间🌹🌹
参考资料
- 1.https://zhuanlan.zhihu.com/p/43927696
- 2.https://github.com/hayashimasa/UNet-PyTorch