QA_全网最系统的单细胞测序细胞互作/细胞通讯分析课程
创始人
2025-05-31 05:08:50
0次
课程介绍见全网最系统的单细胞测序细胞互作/细胞通讯分析课程
本网页的答疑记录来自课程学员群。
文章目录
- 1. 示例数据都提供吗?
- 2. 配套视频在哪里?
- 3. 单细胞预处理
- 4. cellphonedb的环境配置问题
- 5. cellphonedb的标准化问题
- 6. cellphonedb中多个亚基的受配体(complex)如何查询具体的基因?
- 7. 空间转录组绘图
- 8. cellchat中igraph的问题
1. 示例数据都提供吗?
所有代码用到的数据都会提供。
2. 配套视频在哪里?
在B站,UP主:TOP菌
3. 单细胞预处理
-
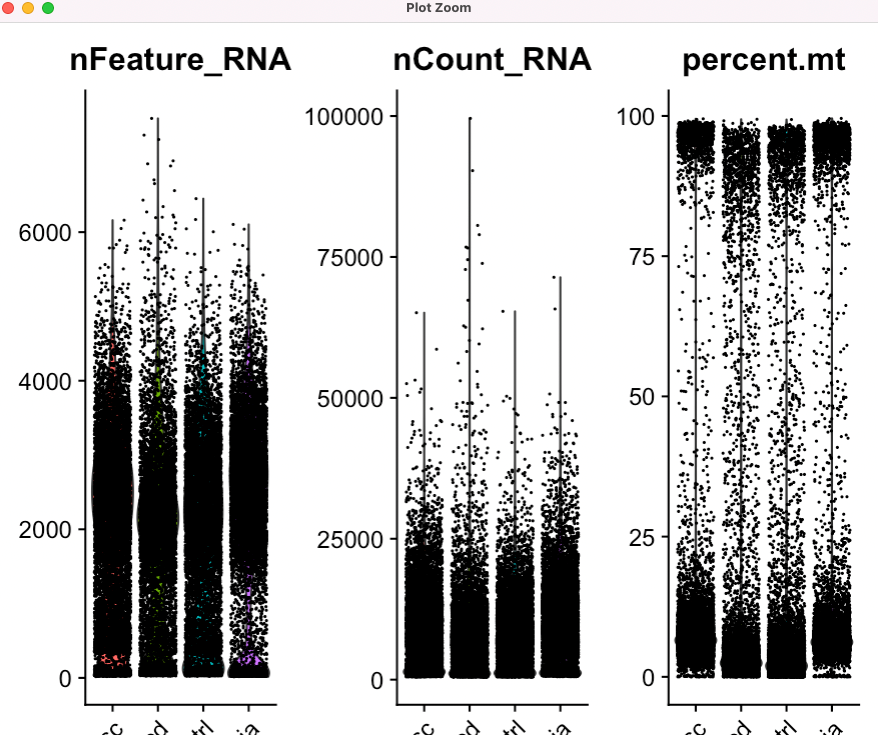
请教一下大家,最右面这个图,4个样本merge后线粒体基因怎么会这么高呀?各自分别跑的时候都没有这么高诶
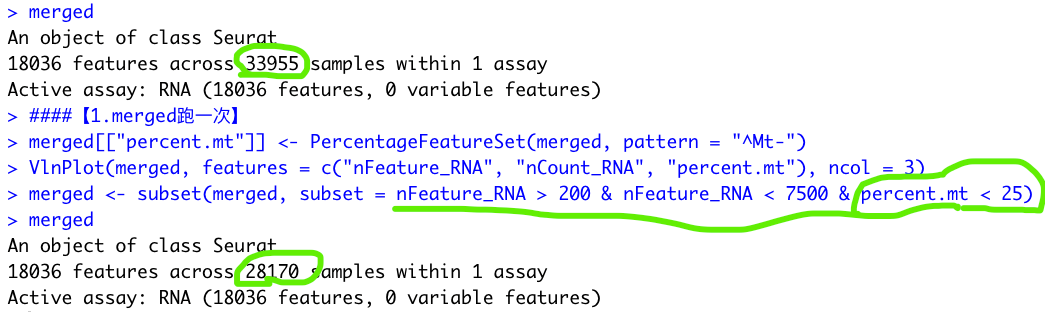
-
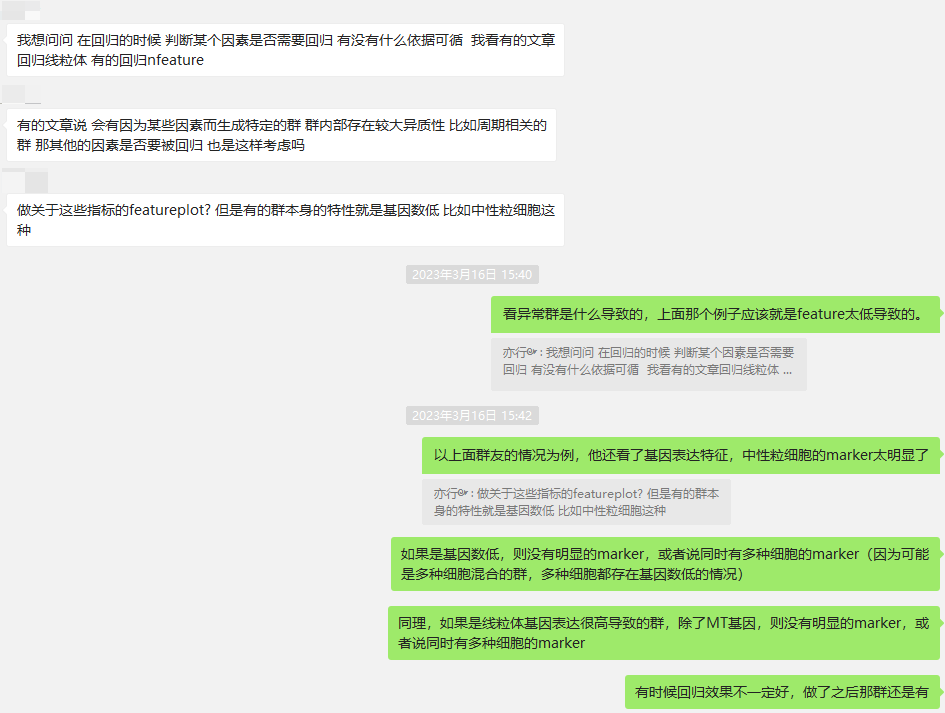
请教大家一个问题,大家质控时,nFeature一般设置多少呀?这群细胞nFeature很低,差异基因很少,cellchat发出和接收的信号都很少,应该在质控时调整cutoff把这群去除嘛?
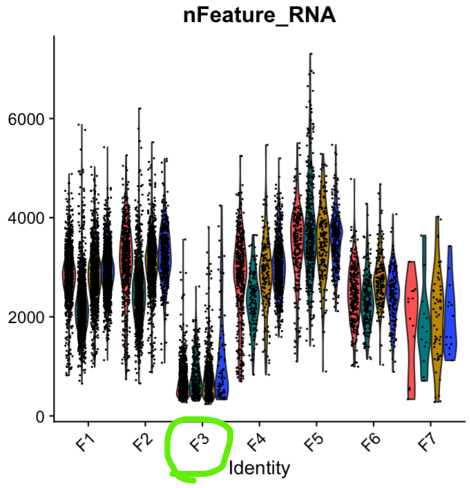
可以的,你的数据feature中位数应该挺高,可以适当增加阈值。可以设成五、六百。然后在scaledata那一步,试一下回归掉基因数,回归与否都试试。
- 我想问问 在回归的时候 判断某个因素是否需要回归 有没有什么依据可循 我看有的文章回归线粒体 有的回归nfeature
4. cellphonedb的环境配置问题
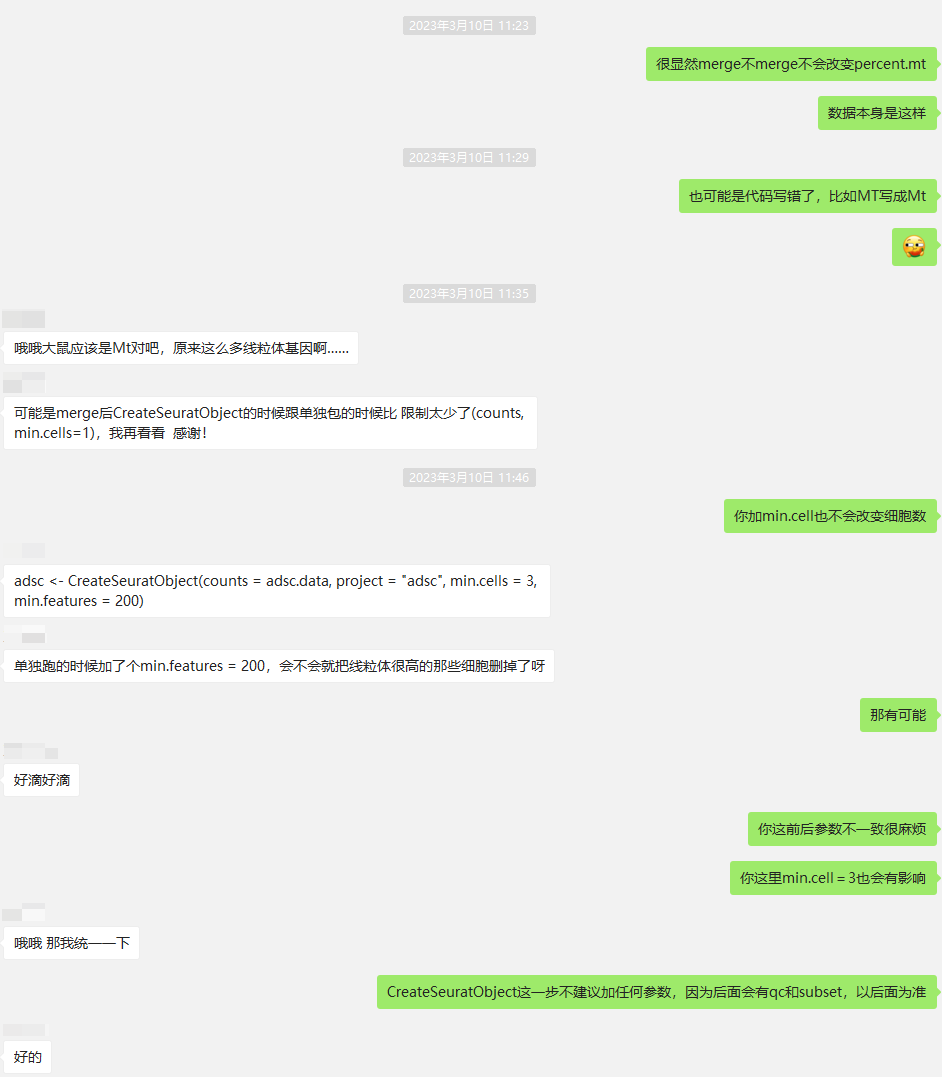
- 求助一个bash脚本运行问题:我把cellphonedb的环境也搭建好了,我单独在terminal中使用conda activate cellphonedb就可以激活环境,然后用bash 运行sh脚本;但是我想学课程的代码把激活环境的代码也放在sh脚本里,直接bash运行sh,然后就运行不了,不知道是什么原因呀
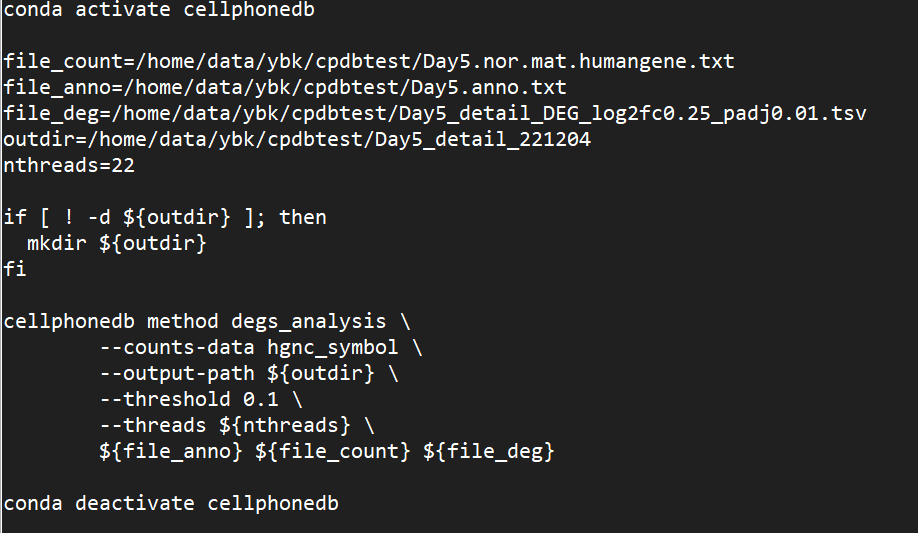
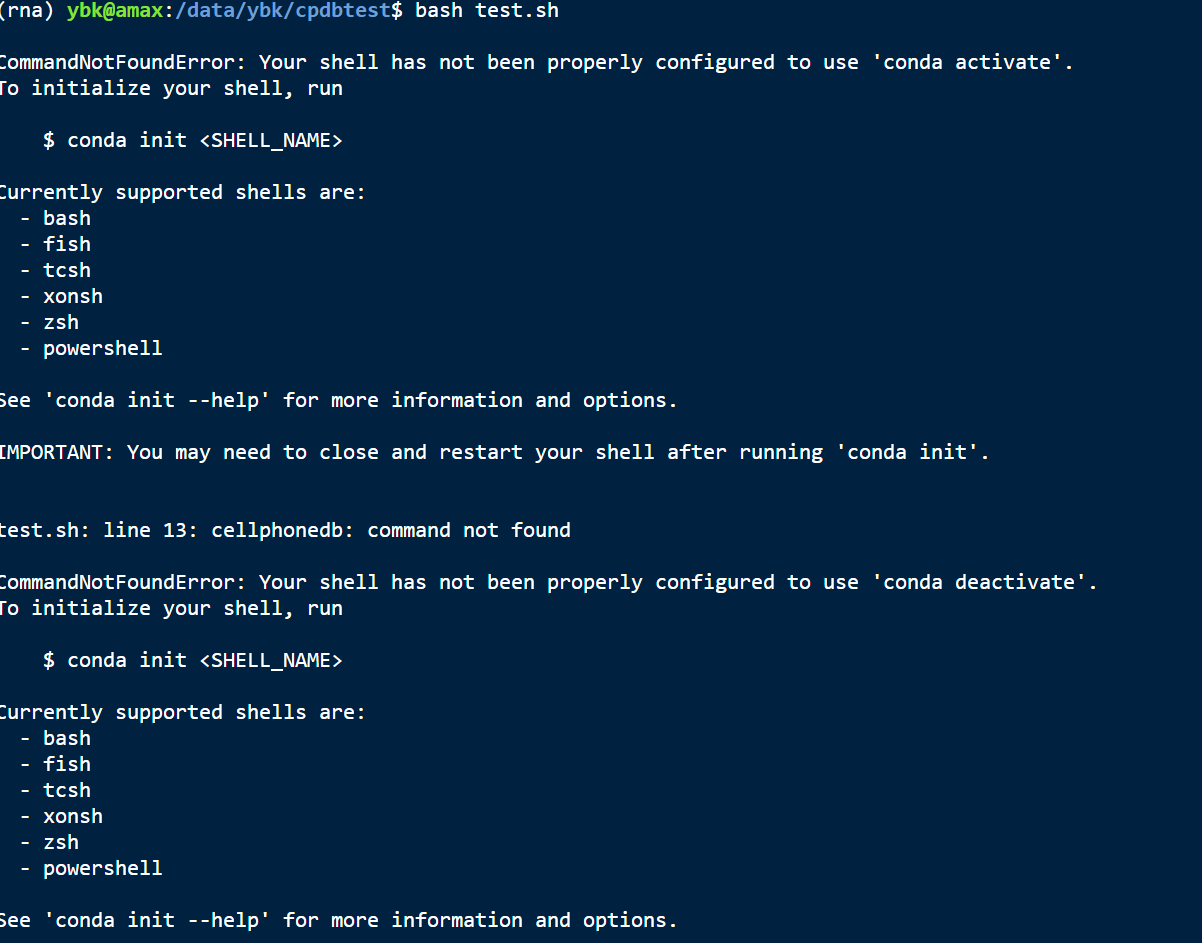
改成下面这种搭配就行了
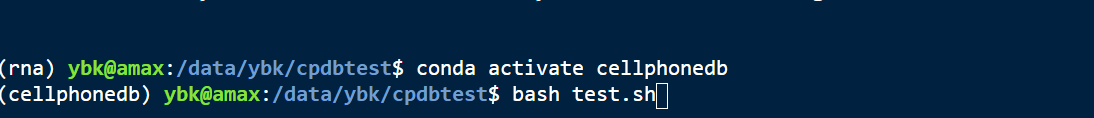
这个问题我不知道原因,也遇到几次。按照你第一种做法,把conda改成source,把activate改成绝对路径(在conda安装包的bin下面),我一般这样改能解决问题
- 想请教一下cellphoneDB安装不上是什么原因呀
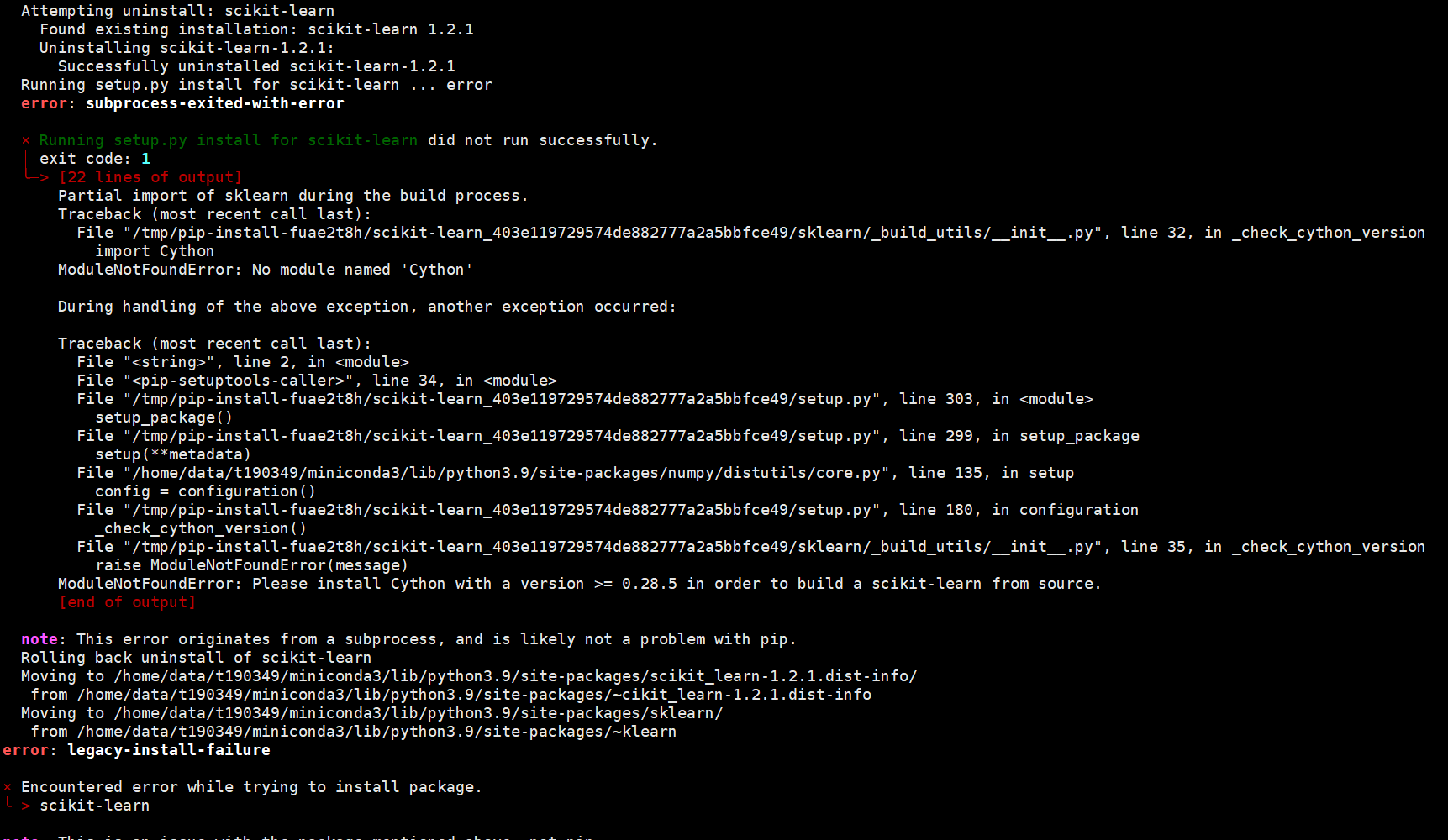
报错是这样
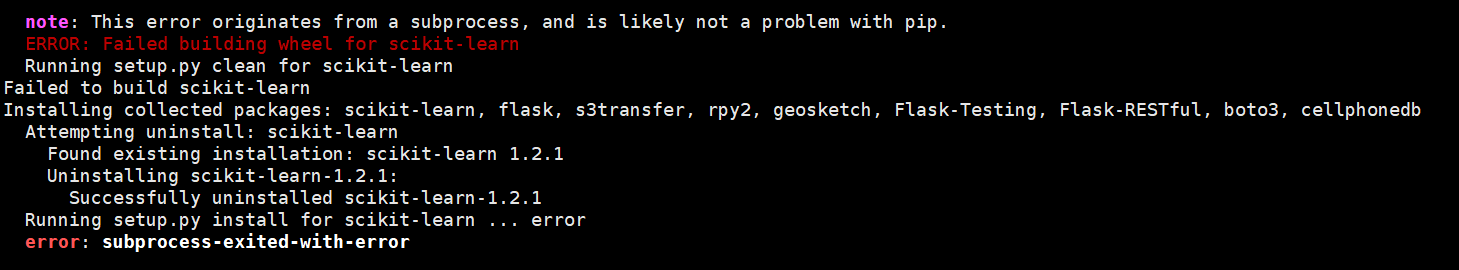
cellphonedb对版本有点严格。按照官网说明,在子环境中下载特定版本的Python。Python需要3.7左右。
- 请问一下,cellphoneDB占用内存大吗?运行久吗?
5. cellphonedb的标准化问题
请问cellphonedb给的数据里有标准化之前的那个矩阵吗?
“1_cpdb测试”文件夹中没有直接提供原始count矩阵; 如果需要的话,“2_cellchat测试a”文件夹中有seurat对象的rds文件,可以尝试提取出count矩阵。
好的,所以是相当于对seurat的count矩阵进行标准化是吧?
是的
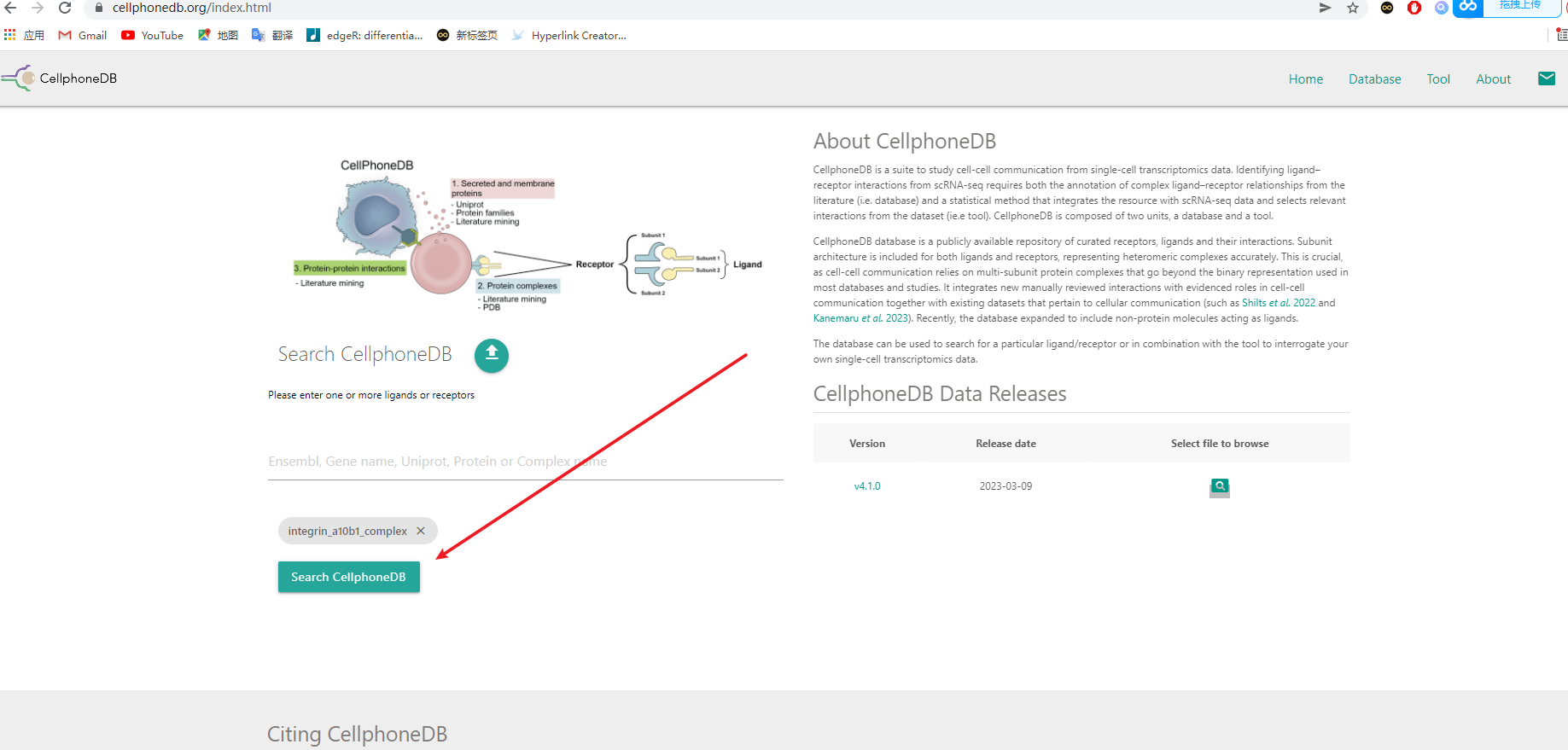
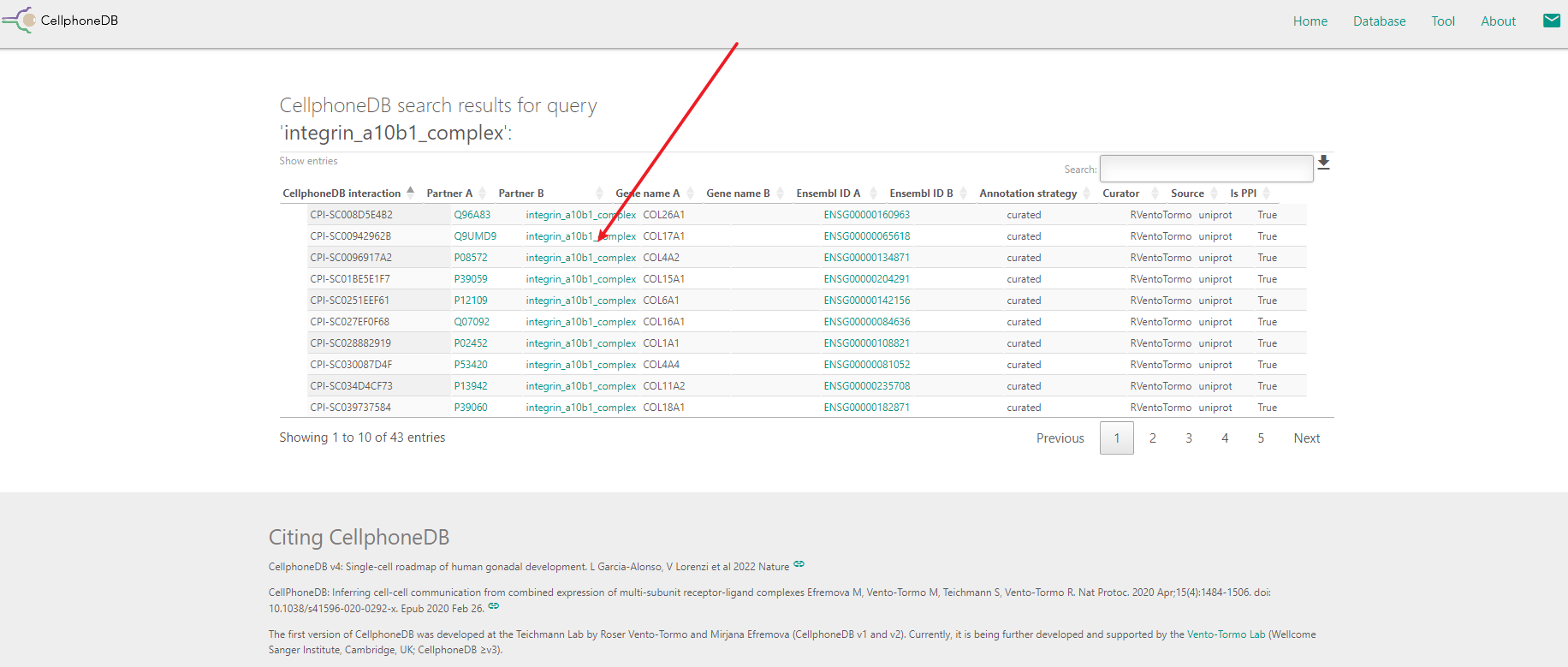
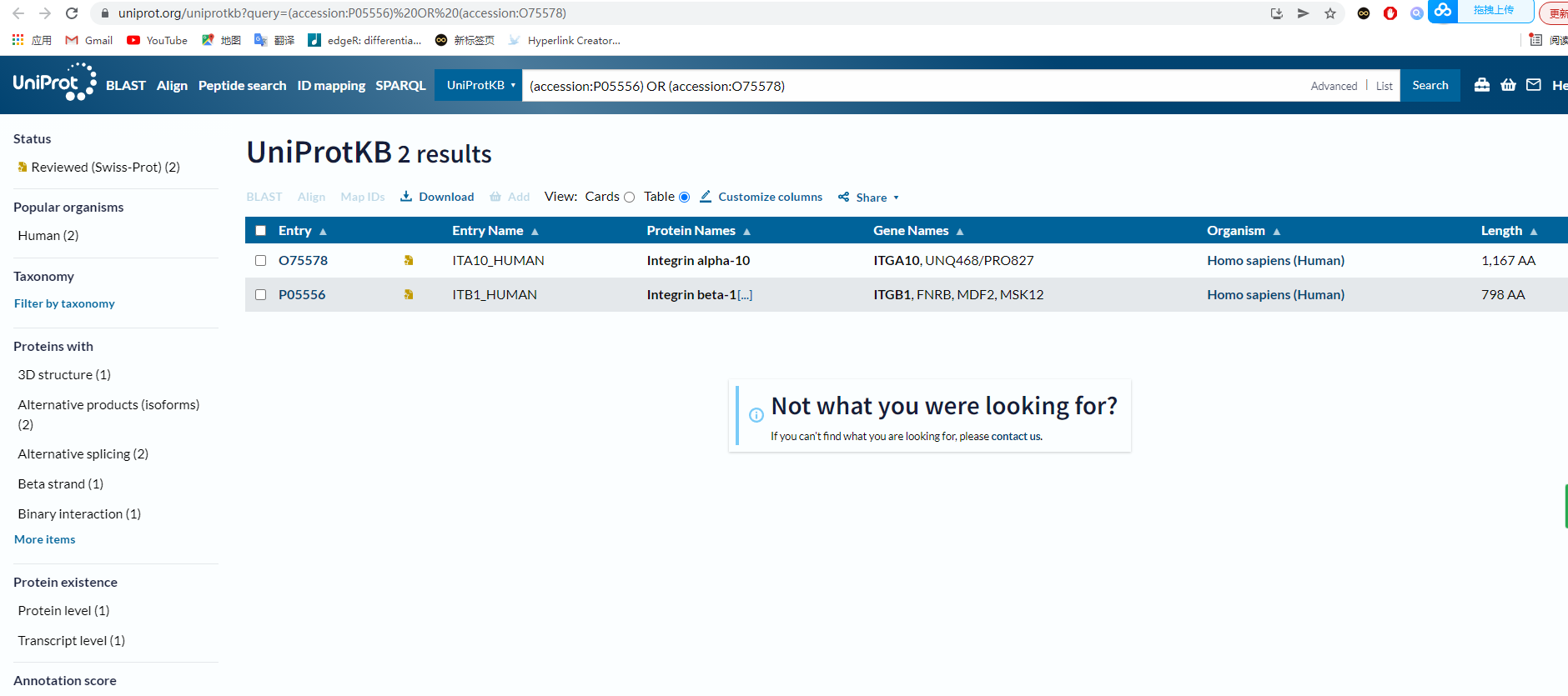
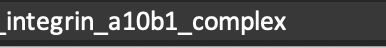
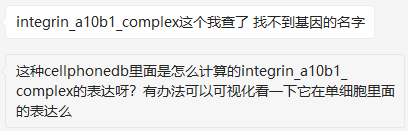
6. cellphonedb中多个亚基的受配体(complex)如何查询具体的基因?
7. 空间转录组绘图
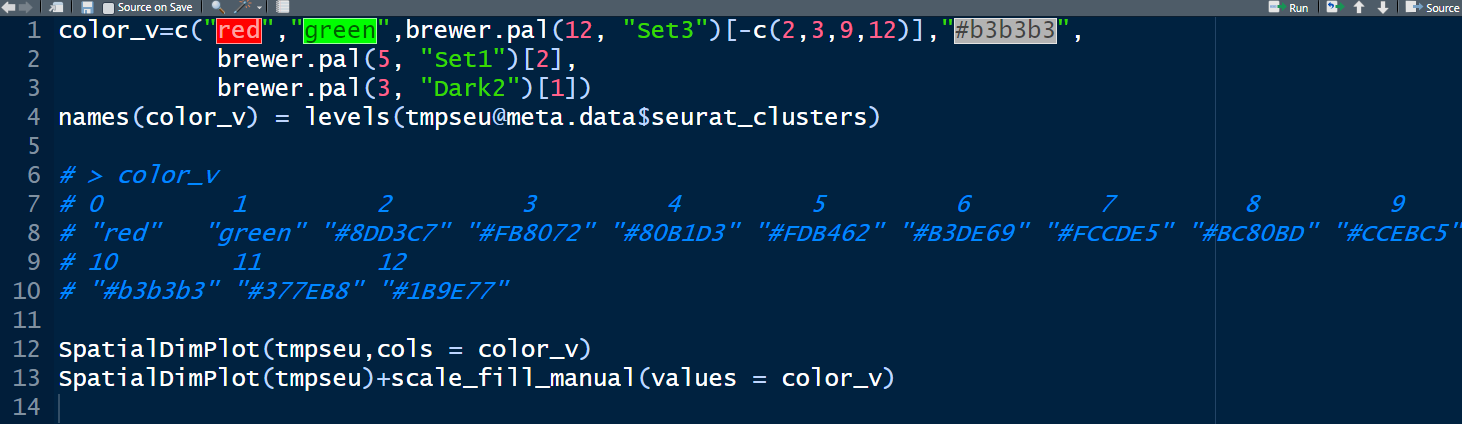
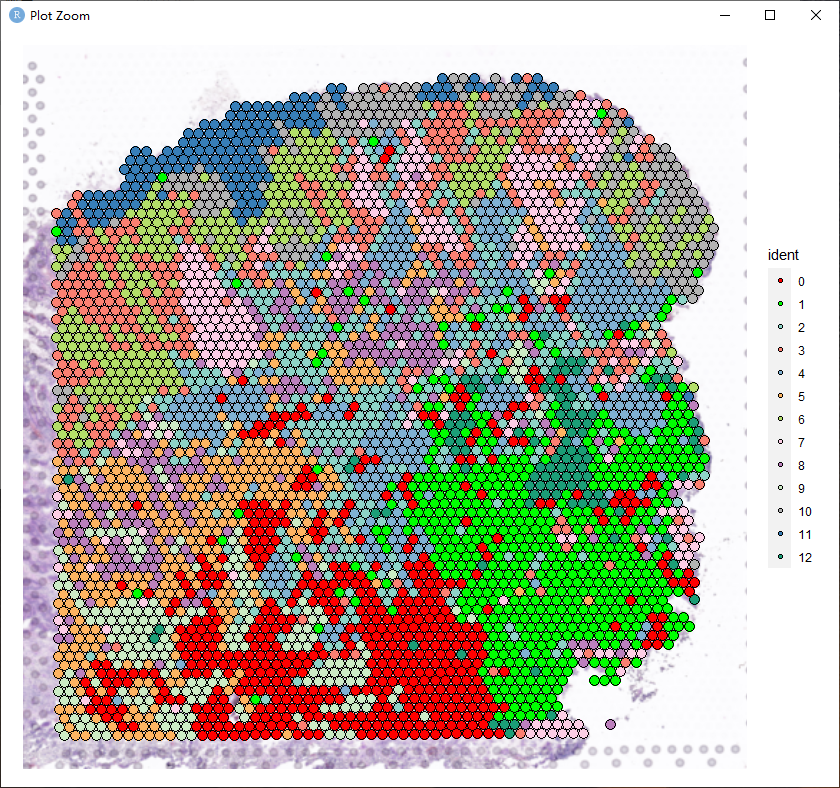
- 请问一下SpatialDimPlot如何修改默认颜色呀?参数cols=修改后画出来还是默认的配色
8. cellchat中igraph的问题
- 请问大家跑cellchat两组比较的代码时,到这几句代码有遇到这个报错的嘛,求问怎么解决的呀?

图中链接(https://blog.csdn.net/x_yAOTU/article/details/124085860)
相关内容
热门资讯
保存时出现了1个错误,导致这篇...
当保存文章时出现错误时,可以通过以下步骤解决问题:查看错误信息:查看错误提示信息可以帮助我们了解具体...
汇川伺服电机位置控制模式参数配...
1. 基本控制参数设置 1)设置位置控制模式 2)绝对值位置线性模...
不能访问光猫的的管理页面
光猫是现代家庭宽带网络的重要组成部分,它可以提供高速稳定的网络连接。但是,有时候我们会遇到不能访问光...
不一致的条件格式
要解决不一致的条件格式问题,可以按照以下步骤进行:确定条件格式的规则:首先,需要明确条件格式的规则是...
本地主机上的图像未显示
问题描述:在本地主机上显示图像时,图像未能正常显示。解决方法:以下是一些可能的解决方法,具体取决于问...
表格列调整大小出现问题
问题描述:表格列调整大小出现问题,无法正常调整列宽。解决方法:检查表格的布局方式是否正确。确保表格使...
表格中数据未显示
当表格中的数据未显示时,可能是由于以下几个原因导致的:HTML代码问题:检查表格的HTML代码是否正...
Android|无法访问或保存...
这个问题可能是由于权限设置不正确导致的。您需要在应用程序清单文件中添加以下代码来请求适当的权限:此外...
【NI Multisim 14...
目录 序言 一、工具栏 🍊1.“标准”工具栏 🍊 2.视图工具...
银河麒麟V10SP1高级服务器...
银河麒麟高级服务器操作系统简介: 银河麒麟高级服务器操作系统V10是针对企业级关键业务...